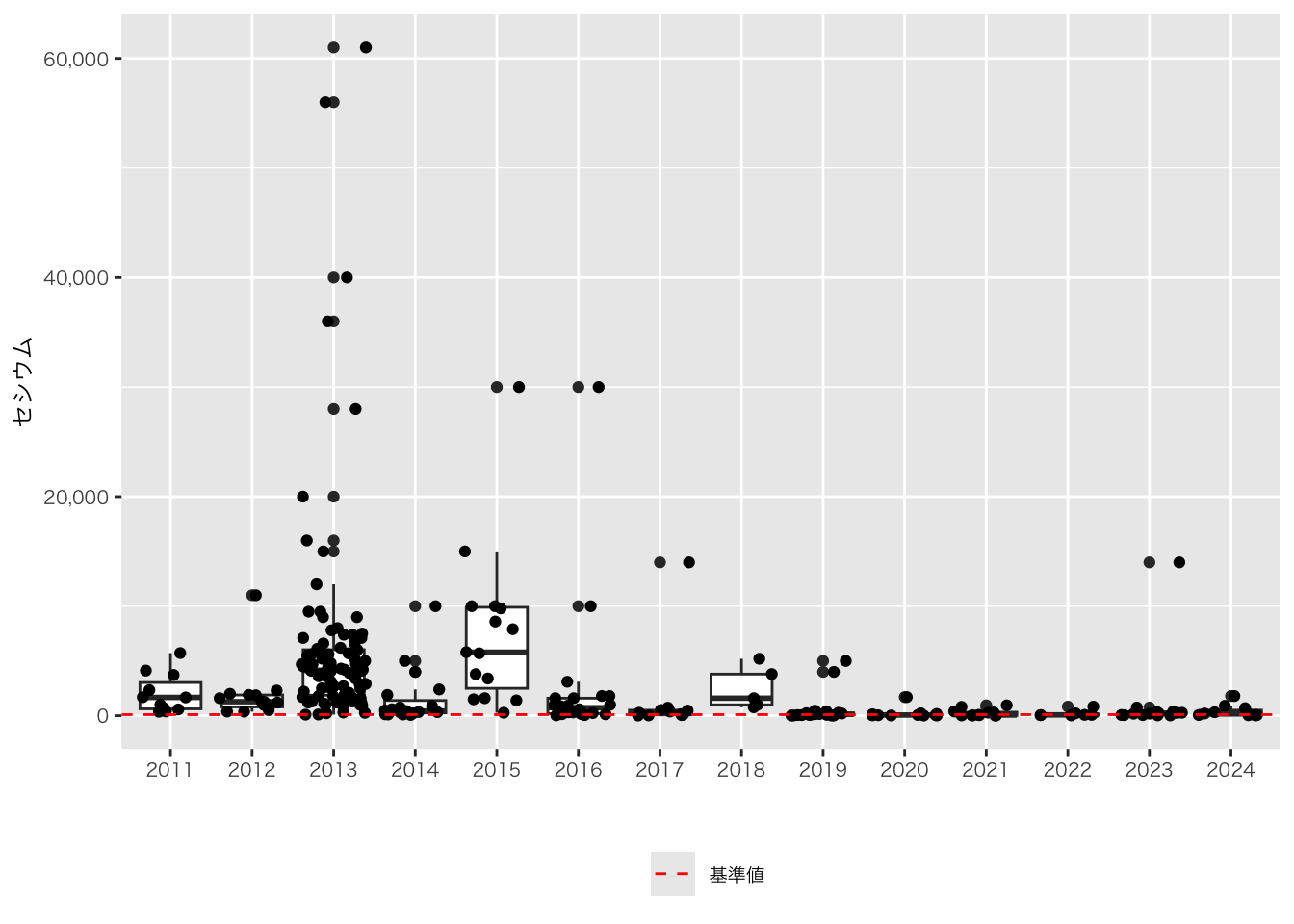
# ggplotのデフォルト設定の調整 ----
## フォントファミリとサイズ
ggplot2::theme_set(
ggplot2::theme_get() +
ggplot2::theme(text = ggplot2::element_text(family = "HiraginoSans-W3", size = 9))
)
## text/labelのフォントファミリとサイズ
ggplot2::update_geom_defaults(
"text",
list(family = "HiraginoSans-W3", size = 3)
)
ggplot2::update_geom_defaults(
"label",
list(family = "HiraginoSans-W3", size = 3)
)
# パッケージの読み込み ----
library(tidyverse) # tidyverse
library(readxl) # エクセルファイルの読み込み
library(skimr) # 統計量の要約
library(scales) # スケール
library(gt) # 表
library(zoo) # rollmean()を使うために必要
library(ggpubr) # ggplot系の追加パッケージ
library(ggrepel) # ggplot系の追加パッケージ
library(gghighlight) # ggplot系の追加パッケージ
library(zipangu) # 元号を西暦に変換するために使用
# データの読み込みと加工 ----
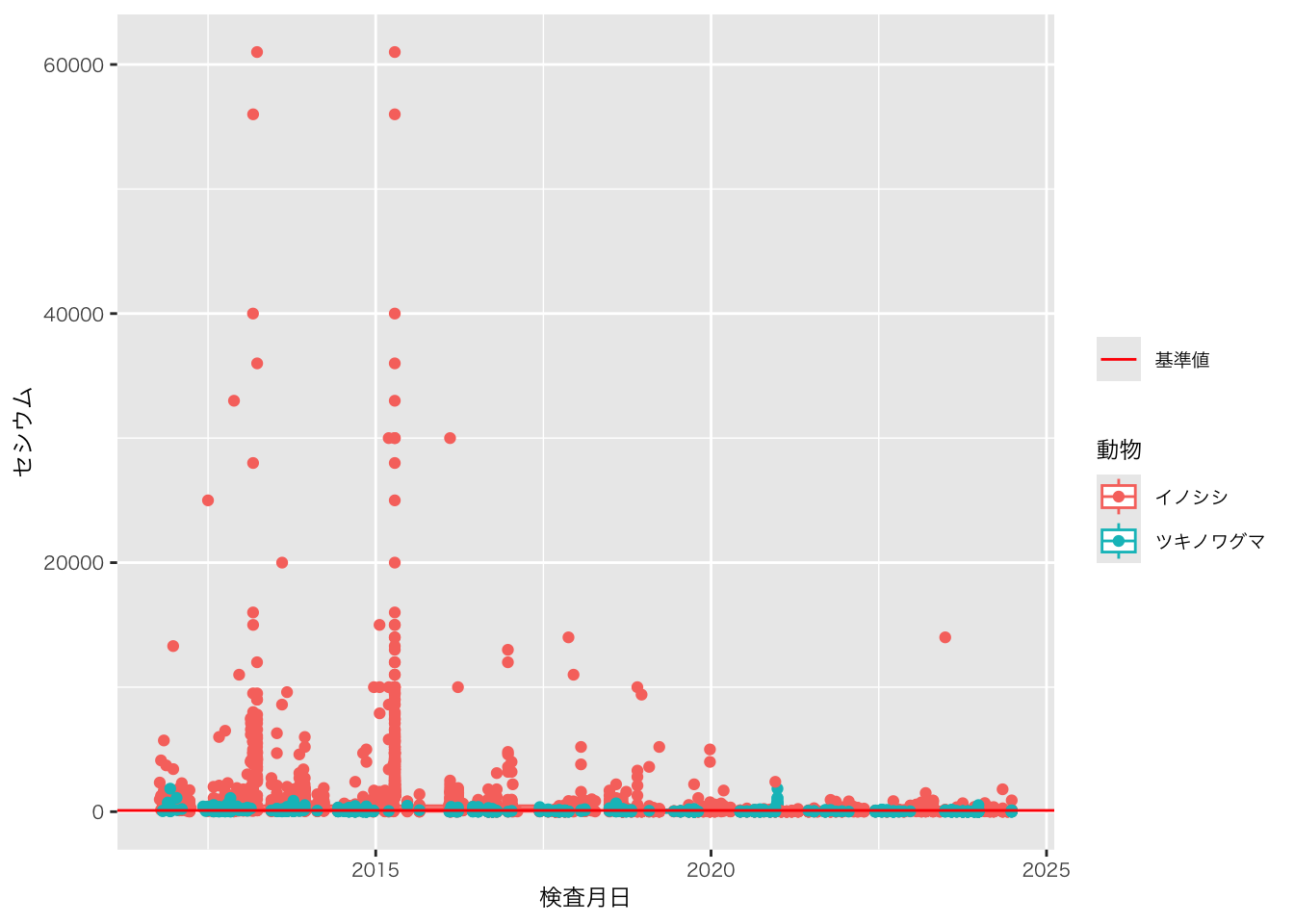
## もともとのデータが動物ごとに作成されている。それぞれの動物ごとにデータフレームを作成し、結合するという手順をとる
df_イノシシ <-
read_csv("data/野生鳥獣の放射線モニタリング調査結果_イノシシ.csv",
# col_names = FALSE,
skip = 1,
locale = locale(encoding = "cp932")) |> #文字コード
slice(-1) |>
slice(1:2320) |>
separate(検査月日, into = c("和暦", "月", "日"), sep = "\\.") |> # 検査月日から年月日を抽出
mutate(
.before = 和暦,
西暦 = convert_jyear(和暦)) |> # 和暦から西暦に変換
select(-5) |>
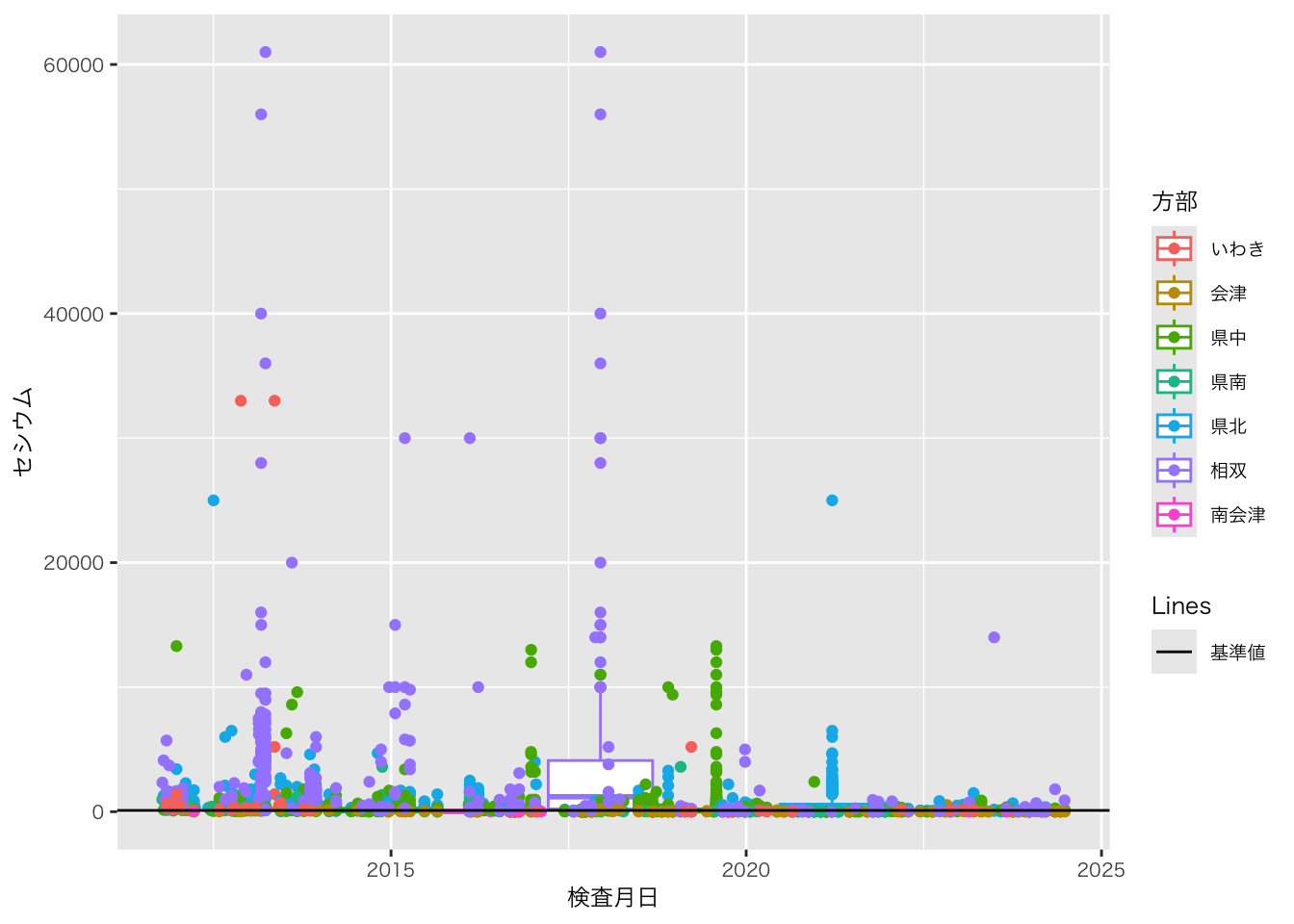
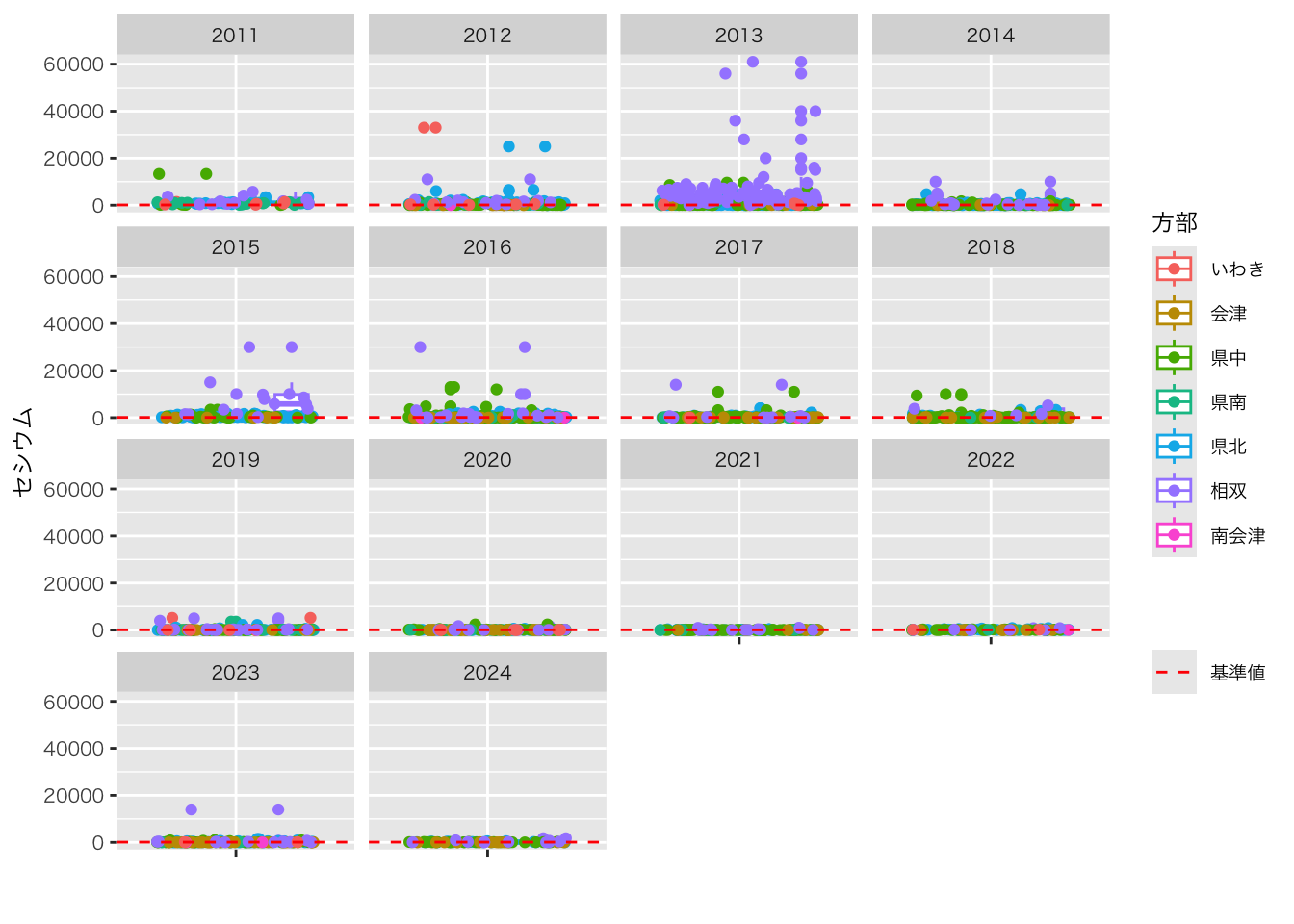
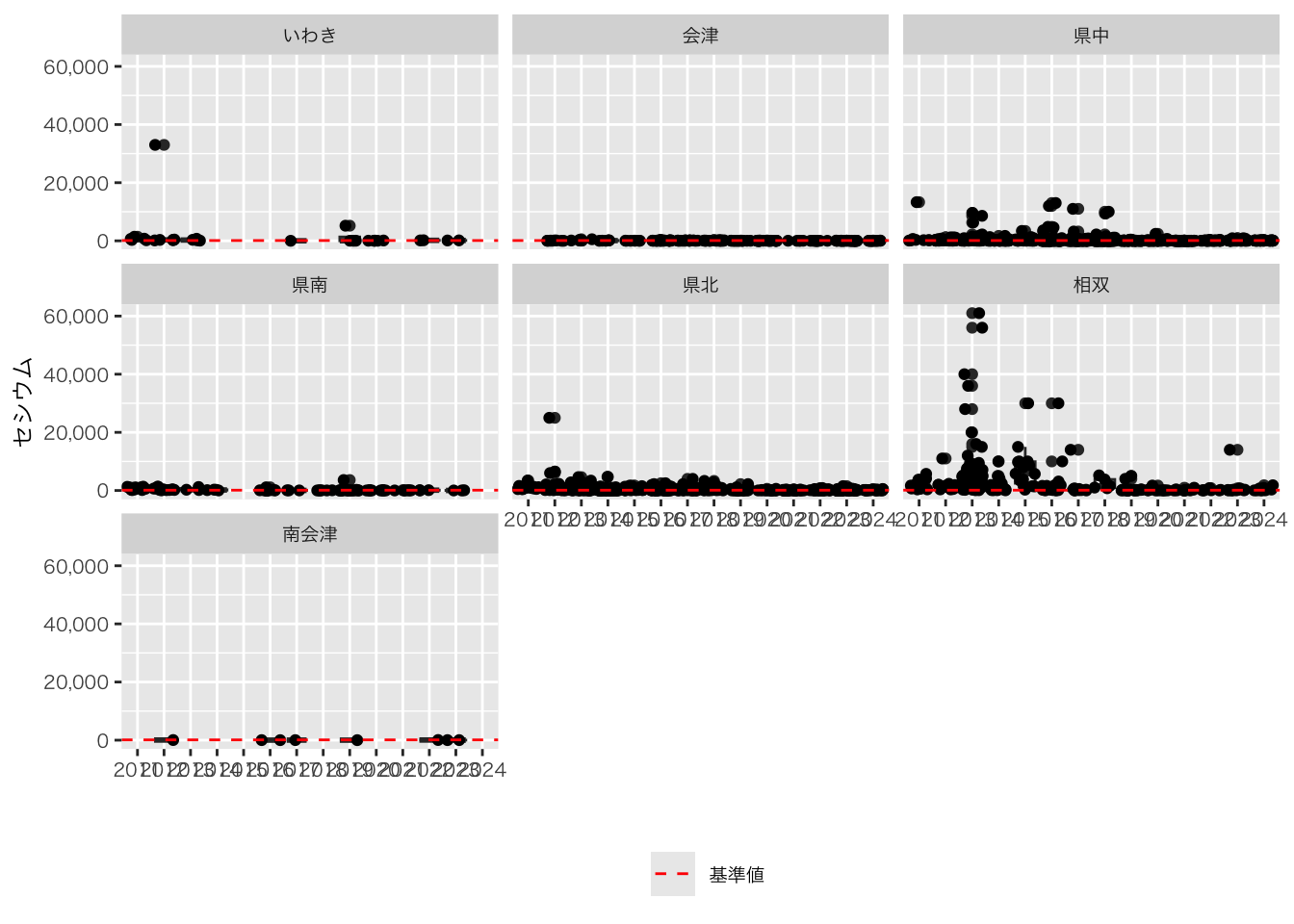
select(No., 方部, 捕獲地点, 西暦, 月, 日, セシウム = `核種濃度\n(セシウム)\nBq/kg`) |>
mutate(
.before = 西暦,
検査月日 = make_date(西暦, 月, 日)
) |>
mutate(
セシウム = as.numeric(gsub(",", "", セシウム))
) |>
mutate(検出有無 = if_else(grepl("検出せず", セシウム), "検出せず", "検出")) |> # 既存のデータから「検出有無」というカラムを作成
mutate(
.after = No.,
動物 = "イノシシ"
)
df_ツキノワグマ <- # 補足については上記を参照
read_csv("data/野生鳥獣の放射線モニタリング調査結果_ツキノワグマ.csv",
# col_names = FALSE,
skip = 1,
locale = locale(encoding = "cp932")) |>
slice(-1) |>
slice(1:813) |>
separate(検査月日, into = c("和暦", "月", "日"), sep = "\\.") |>
mutate(
.before = 和暦,
西暦 = convert_jyear(和暦)) |>
select(-5) |>
select(No., 方部, 捕獲地点, 西暦, 月, 日, セシウム = `核種濃度\n(セシウム)\nBq/kg`) |>
mutate(
.before = 西暦,
検査月日 = make_date(西暦, 月, 日)
) |>
mutate(
セシウム = as.numeric(gsub(",", "", セシウム))
) |>
mutate(検出有無 = if_else(grepl("検出せず", セシウム), "検出せず", "検出")) |>
mutate(
.after = No.,
動物 = "ツキノワグマ"
)
df_野生鳥獣 <- # イノシシのデータフレームとツキノワグマのデータフレームを結合=ggplotに渡すデータフレームを作成
bind_rows(df_イノシシ, df_ツキノワグマ)
df_野生鳥獣 <-
df_野生鳥獣 |>
mutate(
No. = as.numeric(No.),
検査月日 = ymd(検査月日),
# セシウム = as.numeric(セシウム),
月 = as.numeric(月),
日 = as.numeric(日),
動物 = as.factor(動物),
検出有無 = as.factor(検出有無)
)